9편. 효모의 당분 섭취 (4)

구독자 님, 안녕하세요.
저번 편에서는 에일과 라거 효모가 말토오스와 말토트리오스를 얼마나 섭취하는지 비교하며 균주 특성이 당분 섭취에 미치는 영향을 소개해드렸습니다.
이번 Yeasty Letter에서는 효모의 어떠한 특징이 이러한 차이를 가져오는지 구체적으로 알아보려고 합니다.
지난 편을 읽지 않으신 분들은 먼저 읽어보시길 추천드립니다.
앞서 '#7. 효모의 당분 섭취 (2)'에서 언급했듯이, 효모가 말토오스를 섭취하고 분해하기 위해서는 3개의 유전자를 인코딩하는 5가지 MAL loci 중 적어도 하나가 필요합니다. 즉, 이 중 어느 하나라도 갖고 있어야 말토오스와 말토트리오스를 제대로 활용할 수 있다는 뜻이죠.
그렇다면 여러 개의 MAL loci를 보유하고 있으면 말토오스(maltose)와 말토트리오스(maltotriose)를 더욱 효율적으로 사용할 수 있을까요?
이 궁금증을 해소하기 위해, MAL-gene cassettes에 대해 좀 더 살펴보았습니다. 구체적으로는, MAL2 유전자 두 개와 MAL4 유전자 두 개를 갖도록 혼성화(hybridization)한 균주와, 단 하나의 MAL2 유전자만 보유한 균주의 발효 능력을 비교하는 실험을 진행했습니다. 맥아를 이용해 만든 16 Plato 맥즙에서 발효한 결과는 아래 <그림 1>과 같습니다.
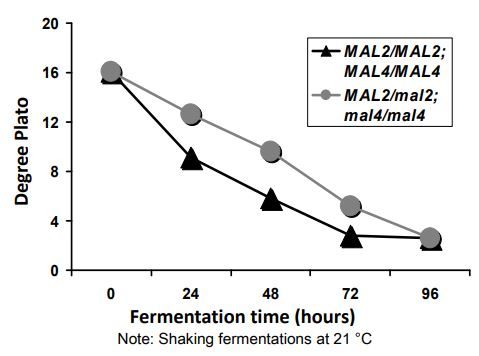
위 그래프에서 삼각형으로 표시된 데이터는 MAL2와 MAL4 유전자를 각각 두 개씩 보유한 효모의 발효 경향을, 동그라미로 표시된 데이터는 단일 MAL2 유전자를 보유한 효모의 발효 경향을 나타냅니다. 그래프를 보면, 여러 MAL 유전자를 가진 균주는 하나의 MAL2 유전자만 보유한 균주보다 전반적인 발효 속도가 더 빨랐습니다. 이는 말토오스를 빠르게 섭취하고 후속 대사 과정을 신속히 진행할 수 있기 때문으로 보입니다.
이처럼 MAL 유전자 서열을 확인하는 것은, 새롭게 발견된 효모가 말토오스와 말토트리오스를 어느 정도까지 섭취하고 발효할 수 있는지 예측하는 데 중요한 근거가 됩니다.
MAL 유전자군 (MAL gene cluster)
- MALS: 말토오스를 포도당으로 분해하는 효소인 α-글루코시다아제를 만드는 유전자
- MALT: 말토오스를 세포 내부로 운반하는 투과효소를 만드는 유전자
- 활성자(Activator): 위 두 유전자의 발현을 함께 조절하는 역할을 합니다.
이 세 유전자가 동시에 존재하거나 복사본이 여러 개인 MAL loci를 보유한 균주는 말토오스와 말토트리오스를 더 효율적으로 운반·분해할 수 있습니다. 투과효소(permease)의 수가 많으면 말토오스나 말토트리오스가 세포 내부로 더 빠르게 유입되고, 말타아제와 같은 가수분해 효소가 충분히 발현되면 해당 당류를 신속히 분해할 수 있기 때문입니다. 또한, 활성자는 이러한 유전자들의 발현 수준을 높여 전반적인 발효 속도와 효율을 향상시킵니다.
이와 같은 분자생물학적 기전을 이해하면, 새로 분리된 효모가 맥주 양조나 기타 발효 공정에서 어떤 성능을 보일지 미리 예측할 수 있습니다. 예를 들어, MAL 유전자 구성에 따라 발효 과정 중 당분 소비 속도, 발효 종료 후 잔류 당의 양, 그리고 최종 알코올 생성량 등을 어느 정도 가늠할 수 있습니다.
나아가, MAL 유전자 서열 분석은 유전자 개량(Genetic Engineering)을 통해 특정 효모 균주의 발효 특성을 향상시키는 데에도 활용될 수 있습니다.
즉, MAL 유전자 서열 확인은 효모가 말토오스와 말토트리오스를 처리하는 기본 역량을 평가하는 핵심 도구로, 발효 산업 전반에서 새로운 균주의 가능성을 예측하고 활용하는 데 큰 도움이 될 수 있습니다.
지금까지 4편에 걸쳐 효모의 당분 섭취에 대해 살펴보았습니다. 주제가 다소 깊어져 부담스러울 수도 있겠지만, 영양 섭취는 그만큼 중요하여 나중에 더 다뤄보고 싶은 욕심도 있습니다.
다음 편부터는 새로운 주제를 가지고 찾아 뵙도록 하겠습니다.
읽어주셔서 감사합니다.
Written by. SB
참고 문헌.
1. Wood, E.J. (1995). "The encyclopedia of molecular biology". Biochemical Education. 23 (2): 1165.
2. Ezkurdia, Iakes; Juan, David; Rodriguez, Jose Manuel; Frankish, Adam; Diekhans, Mark; Harrow, Jennifer; Vazquez, Jesus; Valencia, Alfonso; Tress, Michael L. (2014-11-15). "Multiple evidence strands suggest that there may be as few as 19,000 human protein-coding genes". Human Molecular Genetics. 23 (22): 5866-5878.
3. Sills, A.M.; Stewart, G.G. Production of amylolytic enzymes by several yeast species. J. Inst. Brew. 1982, 88, 313–316.
4. Sills, A.M.; Sauder, M.E.; Stewart, G.G. Isolation and characterization of the amylolytic system of Schwanniomyces castellii. J. Inst. Brew. 1984, 90, 311–314
5. Stewart, Graham G. Brewing and Distilling Yeasts. Springer International Publishing, 2017.
6. Bisson, L.F.; Coors, D.M.; Frankel, A.L.; Lewis, D.A. Yeast sugar transporters. CRC Crit. Rev. Biochem. Mol. Biol. 1993, 284, 259–308.
7. Zheng, X.; D’Amore, T.; Russell, I.; Stewart, G.G. Factors influencing maltotriose utilisation during brewery wort fermentations. J. Am. Soc. Brew. Chem. 1994, 52, 41–47.
8. Russell, I.; Stewart, G.G. Transformation of maltotriose uptake ability into a haploid strain of Saccharomyces spp. J. Inst. Brew. 1980, 86, 55–59.
9. Sherman, F.; Fink, G.R.; Lawrence, C.W. Methods in Yeast Genetics; Cold Spring Harbour Laboratory: Cold Spring Harbor, NY, USA, 1979.










의견을 남겨주세요